Konrad-Zuse-Schule beteiligt sich am Kampf gegen das SARS-CoV-2 Virus und setzt freie Rechnerkapazitäten zur Viren-Forschung ein
Die Konrad-Zuse-Schule beteiligt sich am Folding@home Projekt und setzt ihre, zurzeit freien, Rechenkapazitäten im Kampf gegen das SARS-CoV-2 Virus ein. Über das von der Stanford-University in Leben gerufene Projekt „Folding@home“ ist es möglich, die Rechenleistung gewinnbringend einzusetzen.
Jens Heddrich, IT-Lehrkraft an der Konrad-Zuse-Schule erläutert die Idee wie folgt: „Trotz der Ausgangsbeschränkungen und dem Kontaktverbot ist es über das Internet möglich, einen kleinen Beitrag zur Bekämpfung des Corona-Virus zu leisten. Dies geht sogar über Nacht während man schläft. Während unsere Server derzeit auf Hochtouren laufen, um gemeinsames Lernen über unsere Plattformen zu ermöglichen, sind die Rechner der Schule derzeit "arbeitslos". Einem Aufruf der Universität Stanford folgend, werden unsere Rechnerkapazitäten für die Dauer der Schulschließung daher am Projekt Folding@home teilnehmen, mit dem computergenerierte Modelle von Proteinstrukturen erstellt werden, die bei der Forschung gegen das Corona-Virus helfen sollen“.
Im März 2020 startete die Stanford University in den Vereinigten Staaten das Programm zur Unterstützung von Forschern auf der ganzen Welt. Die Community arbeiten daran, ein Heilmittel zu finden und mehr über den Ausbruch von COVID-19 im Rahmen des Volunteer-Computing-Projektes Folding@home, zu erfahren. Das Projekt Folding@home der Stanford University wurde 2000 ins Leben gerufen und hat das Ziel der Erforschung von Krankheiten, in dem die Proteinfaltung und andere Arten von Molekulardynamiken simuliert wird.
Statt die Rechenleistung eines einzelnen Rechners zu nutzen, wird dabei eine komplexe Aufgabe in Teilaufgaben aufgeteilt, diese auf mehrere Rechner verteilt und deren Rechenleistungen zur Aufgabenbewältigung genutzt. Das Projekt nutzt durch verteiltes Rechnen die ungenutzten Verarbeitungsressourcen von Personalcomputern und Servern auf denen die Software installiert ist und die so zur Erforschung von Krankheiten beitragen.
Das Projekt verwendet hierbei eine statistische Simulationsmethode, die gegenüber traditionellen Berechnungsmethoden einen Paradigmenwechsel darstellt. Als Teil des Client-Server-Modells erhalten die Teilnehmer (Clienten) nach Anforderung eines Dienstes beim Server jeweils Teile einer Simulation (Arbeitseinheiten/Work-Units), berechnen und vervollständigen sie und geben sie an die Datenbankserver des Projekts zurück, wo die Einheiten sodann zu einer Gesamtsimulation zusammengestellt werden.
Der Hauptzweck des Projekts ist die Bestimmung der Mechanismen der Proteinfaltung, d. h. des Prozesses, durch den Proteine ihre endgültige dreidimensionale Struktur erreichen, und die Untersuchung der Ursachen von Proteinfehlfaltungen. Dies ist von Interesse für die medizinische Forschung über SARS-CoV-2, Alzheimer, Huntington und viele Formen von Krebs, neben weiteren, anderen Krankheiten. In geringerem Umfang versucht Folding@home auch, die endgültige Struktur eines Proteins vorherzusagen und zu bestimmen, wie andere Moleküle mit ihm interagieren können, was sich auf die Entwicklung von Medikamenten auswirkt.
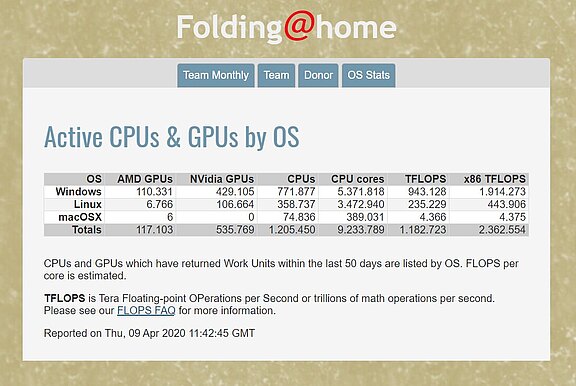
Folding@home: 1 Million Systeme rechnen mit 2,3 ExaFLOPS gegen COVID-19
Auf der Suche nach Lösungen gegen die vom Coronavirus ausgelöste Krankheit COVID-19 wächst das Engagement für Volunteer-Computing-Projekte weiter. Folding@home hat Anfang April die Marke von über einer Million Systeme geknackt und kommt inzwischen auf eine kombinierte Rechenleistung von mehr als 2,3 ExaFLOPS.
Das Distributed-Computing-Projekt hat dank Coronavirus mehr Rechenkapazität als aktuelle Supercomputer und vorerst einen neuen Rekord aufgestellt:
„Wir hoffen mit unseren Möglichkeiten, dem Engagement der Kolleginnen und Kollegen und der zur Verfügung stehenden Hardware ein wenig beim Kampf gegen das CORONA-Virus beitragen zu können. Dem Landkreis Fulda danken wir für die Unterstützung, dass die technische Ausstattung für diesen Zweck genutzt werden darf“, so Schulleiterin der Konrad-Zuse-Schule Hünfeld, Susanne Diegelmann abschließend.
Zusatzinformationen
Detaillierte Darstellung der Vorgehensweise im Kampf gegen das COVID-19 VirusProteine sind die absoluten Alleskönner der Zellen.
Proteine, auch Eiweiße genannt, sind Biomoleküle, die aus Aminosäuren bestehen. Proteine sind von zentraler Bedeutung für Organismen. So sind sie wesentlicher Bestandteil von Biomembranen und katalysieren als Enzyme sämtliche Stoffwechselreaktionen in Lebewesen. Außerdem übernehmen sie weitere lebenswichtige Aufgaben wie Bewegung mithilfe von Muskeln, Abwehr von Krankheitserregern, Stabilität durch Gerüstproteine in Knochen, Knorpel und Bindegewebe sowie den Transport von Sauerstoff mittels Hämoglobin, dem roten Blutfarbstoff.
Proteine werden zu komplexen 3D-Strukturen gefaltet.
Doch wie können Proteine so vielfältige Aufgaben übernehmen? Werden Proteine im Körper benötigt, werden entsprechende Bereiche der DNA, dem Träger der Erbinformation, im Zellkern „kopiert“ und dann im Zellplasma in eine Abfolge von Aminosäuren übersetzt. Dabei entsteht, vergleichbar mit einer Perlenkette, eine lange Kette aus Aminosäuren. Um ihre entsprechende Funktion zu erfüllen, müssen die Proteine aber erst noch ihre räumliche Struktur einnehmen. Dieser als Proteinfaltung bezeichnete Prozess ist aufgrund der Vielzahl möglicher Faltungen, die mit der Länge der Aminosäurekette exponentiell zunehmen, extrem kompliziert. Erst durch die Proteinfaltung erhalten Proteine ihre spezifische dreidimensionale Struktur, die die Voraussetzung für eine fehlerfreie Funktion darstellt.
Fehl-Faltungen haben verheerende Folgen.
Doch was hat nun die Proteinfaltung mit Krankheiten bzw. deren Therapie zu tun? Wissenschaftler gehen davon aus, dass Krankheiten wie Alzheimer, Rinderwahnsinn, Chorea Huntington, cystische Fibrose, aber auch einige Krebsarten das Resultat falscher Proteinfaltung sind, wodurch diese Proteine ihre Funktion verlieren oder aber miteinander verklumpen und sich z.B. im Gehirn ansammeln und so entsprechende Symptome auslösen können.
Großcomputer erforschen durch Simulation die Vorgänge, die zu Fehl-Faltungen führen.
Die Erforschung solcher Proteinfehlfaltungen war lange Zeit aufgrund der unzähligen Faltungsmöglichkeiten einer Aminosäurekette nur schwer möglich. Dank der enormen Rechenleistung heutiger Großcomputer besteht inzwischen jedoch die Möglichkeit, Simulationen von Proteinfaltungen am Computer durchzuführen. Dies nutzt das Projekt Folding@home, um Proteinfehlfaltungen näher zu untersuchen und die Entwicklung von Arzneimitteln voranzutreiben.
Das COVID-19-Pandemie-Virus könnte durch medikamenteninduzierte Fehlfaltung gestoppt werden.
Derzeit unterstützt das Projekt Folding@home Forscher auf der ganzen Welt bei der medizinischen Erforschung des Coronavirus Sars-CoV-2. Ziel ist es, zu verhindern, dass das Virus Zellen erkennen und in diese eindringen kann. Ebenso wie alle Viren besitzt auch Sars-CoV-2 Proteine, die es dem Virus ermöglichen, sich innerhalb einer Zelle selbst zu vervielfältigen, aber auch das Immunsystem der infizierten Person zu unterdrücken und so der Immunabwehr zu entgehen. Mit computerbasierten Simulationen der Faltung der Sars-CoV-2-Proteine zielt Folding@home darauf ab, Medikamente zu entwickeln, die das Virus stoppen, indem sie z.B. an bestimmten Stellen von Zielproteinen binden und so deren Funktion hemmen. Diese Simulationen sollen dabei helfen, genau solche Stellen zu identifizieren. Im Labor kann dann anschließend das Design von Medikamenten beginnen, die an die gefundene Proteinstelle binden und so eine Infektion verhindern.
Podcast
Zuse-Schule Hünfeld unterstützt US-Forscher bei der Suche nach Corona-Schwachstellen (15:30)Autor: Thomas Kurella
Veröffentlicht am 23.04.20 um 14:05 Uhr
Quelle: © Hessischer Rundfunk